目前我正在做recon-all,后续怎么使用AAL模版来代替aparc模版提取信息呀,是将它的nii格式想办法转换为annot,然后再挨个提取嘛?感觉有点迷茫 ![]()
![]()
![]()
- 基本思路就是将AAL转换为annot文件,可以参考以前的讨论:https://brainbbs.cn/t/topic/368
- 不过这种转换是不完美的,因为在volume空间连续的脑区转到surface上可能就不连续了,一般需要后续编辑。如果不能在网上找到现成AAL的annot文件,这个工作量感觉还是比较大。
- 为什么要选择用AAL分区呢?感觉这个分区从准确性上来说很不好。
感谢Alex老师,我看完了,收获很多 ![]()
![]()
![]() ,使用AAL分区是老师的要求,我现在准备尝试在网上搜索一下看有没有现成的
,使用AAL分区是老师的要求,我现在准备尝试在网上搜索一下看有没有现成的
网上的这个AAL2的annot文件我以前看过,确实没有标签信息(也就是每个编号表示哪个脑区)。如果是我的话,我会把这个annot文件转换到MNI空间,然后和AAL的MNI模板进行匹配,看看annot文件里的编号对应于AAL模板里的哪个编号和脑区。不知道我说清楚了没?如果有什么不清楚的地方,我们再讨论。
非常非常感谢Alex老师,我基本懂您的意思了,我再去研究一下怎么实现 ![]()
![]()
![]()
Alex老师好,最近被一些事情耽搁了,我对神经影像相关的内容了解还不太深,最近也看了您写的一些关于MNI152和fsaverage相互转换,以及对MNI空间与模板的解读。
目前我的想法就是按照您之前那篇文章,将fsaverage模板的脑网络(aal.annot)转换到Colin27空间中,我比较疑惑的就是之前看到您的有篇帖子有写AAL模板是Colin27,不是MNI152空间,也就是存在不同的MNI空间,并且Colin27在不同的地方使用的内容都不同,我该如何在freesurfer中使用它呢,或者说使用哪个版本(不知道我有没有理解错误),对应您之前文章的代码,也就是这两条
‘/home/Alex/Wu2017_RegistrationFusion/bin/final_warps_FS5.3/allSub_fsaverage_to
_FSL_MNI152_FS4.5.0_RF_ANTs_avgMapping.prop.mat’, …
‘/home/Alex/Wu2017_RegistrationFusion/bin/liberal_cortex_masks_FS5.3/FSL_MNI152
_FS4.5.0_cortex_estimate.nii.gz’);
麻烦老师了
- 我博客里的这个代码用的是MNI152的文件,因为我希望转换到MNI152的空间上。你如果需要转换到Colin27的模板空间,RegistrationFusion也提供了对应的文件,就是把上面代码里的
FSL_MNI152换成SPM_Colin27就行了。 - 如果你只是想知道annot文件里的脑区标签,我估计MNI152和Colin27差别很小。
好的老师 ![]()
![]()
![]() ,非常感谢,我继续研究后面的步骤
,非常感谢,我继续研究后面的步骤
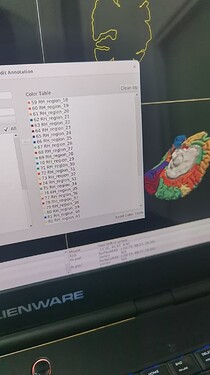
老师晚上好,打扰了,我现在成功提取了转化后的文件,但是我还是不知道有什么方法可以去将这两个模板进行匹配对比 ![]()
![]()
![]() 。我查询资料思考后的想法是先分别读取两个文件,发现ALL2(称为aal.nii)是91x109x91 ,而转化后的AAL文件(annot.nii)是256x256x256,两者维度不匹配,然后通过重采样,使得annot变为91x109x91 ,再读取他们的标签信息。
。我查询资料思考后的想法是先分别读取两个文件,发现ALL2(称为aal.nii)是91x109x91 ,而转化后的AAL文件(annot.nii)是256x256x256,两者维度不匹配,然后通过重采样,使得annot变为91x109x91 ,再读取他们的标签信息。
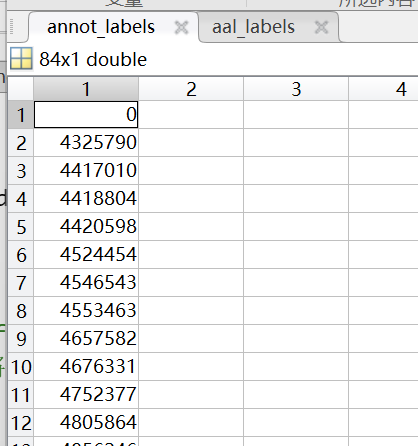
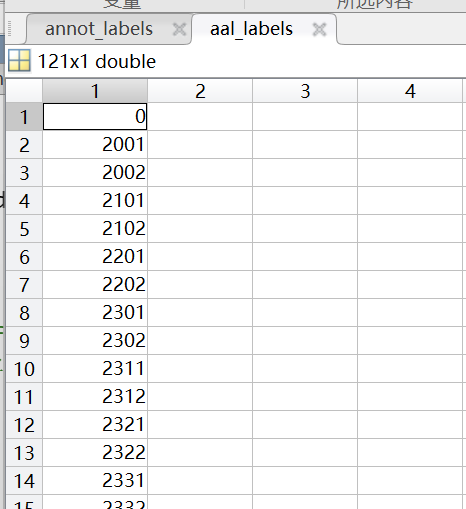
但是之后我就不太理解了,我发现annot的label是84x1 的,而aal的label是121x1的,如何去对比匹配呢?而且我对里面的数值代表什么也不太理解 ![]()
![]()
![]() 下面是两者读取出来的label的截图,麻烦Alex老师了
下面是两者读取出来的label的截图,麻烦Alex老师了
- 首先annot只包括皮层脑区,aal的120个脑区还包括皮下核团和小脑,所以匹配前应只保留皮层的部分。
- 因为annot文件格式比较特殊(参考官方wiki),所以RegistrationFusion转换完成后每个体素的值是annotation value,也就是说这个值虽然也是每个脑区不同,但是并不是每个脑区的编号。annotation value和脑区的对应关系在
colortable.table里可以找到,第5列是annotation value。所以在保存结果之前还需要对结果进行一次转换。 - RegistrationFusion转换后的空间是FreeSurfer的conformed space,所以维度不一样。可以用下面的代码再进行一个变换:
mri_label2vol --seg annot.nii.gz --temp colin27.nii.gz --o output.nii.gz --regheader annot.nii.gz
好的Alex老师,不好意思,今天课有点多,没能及时回复,我现在正在阅读这个官方文档,非常感谢老师的解答和帮助 ![]()
![]()
![]()
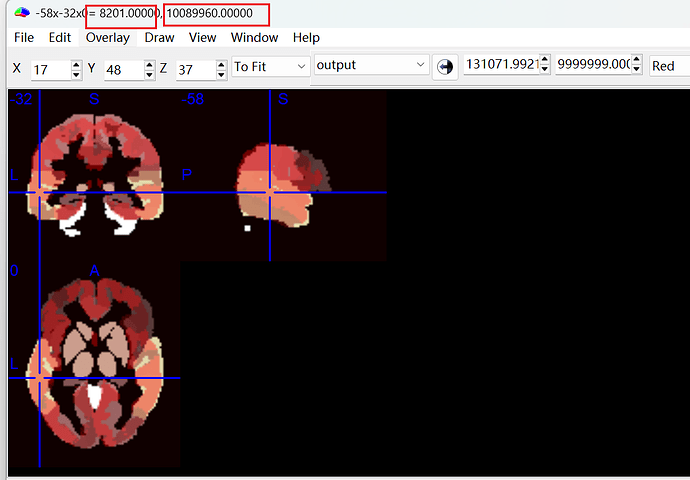
Alex老师晚上好,目前我完成了您所说的变换,下面是我关于匹配对比的想法:我准备使用mricon里的overlay进行对比,图片里前面的8201是AAL2模板里的Temporal_Mid_L,后面的10089960对应的是 LH_region_39,逐一对比找出每个值对应的脑区,这样可行吗,感谢Alex老师
我觉得这个思路没问题。
Alex老师 ![]()
![]()
![]() 我在这么对比的时候发现有的地方不是一一对应关系是什么原因以及如何处理呀
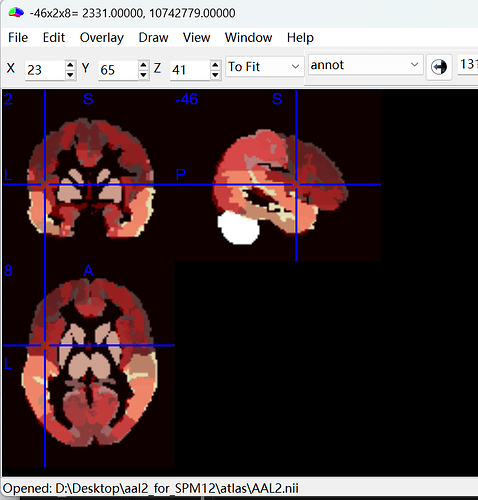
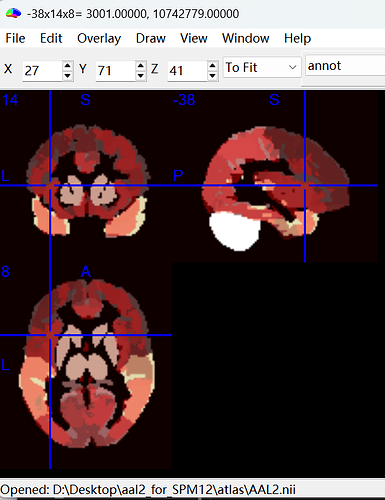
我在这么对比的时候发现有的地方不是一一对应关系是什么原因以及如何处理呀 ![]() ,比如说在这里10742779对应的’LH_region_16’,既对应33 Insula_L 3001,也对应13 Rolandic_Oper_L 2331,而之前的对比里还发现13 Rolandic_Oper_L 2331是对应’LH_region_7’,麻烦了老师
,比如说在这里10742779对应的’LH_region_16’,既对应33 Insula_L 3001,也对应13 Rolandic_Oper_L 2331,而之前的对比里还发现13 Rolandic_Oper_L 2331是对应’LH_region_7’,麻烦了老师
这种情况很正常,因为在边界的地方是有一些不确定性,因为你是从annot转到MNI的,所以和原始的AAL不是完全对应的。一个量化的方法就是算一下重合比例,比如LH_region_16和Insula_L重合率为10%,与Rolandic_Oper_L重合率为80%,那么LH_region_16应该就是对应Rolandic_Oper_L。这个问题在匹配不同分区模板的时候也会出现,在前面的帖子1、帖子2中讨论过。
谢谢Alex老师 ![]() ,我现在就去看看
,我现在就去看看
Alex老师早上好!我又带着问题来打扰了 ![]()
- 我在计算Dice相关系数时,使用120个脑区的aal2文件和83个脑区(包含82个脑区和脑区???,应该就是背景部分吧)的annot文件,但是发现生成的文件是121x84的,aal2模板里多出的那个应该就是aal2对应的0的背景部分,但是annot文件又多出来一个label 0 ,变成了84个
 请问对结果有影响吗,我能否直接把dice生成的文件里对应得行列直接删掉。
请问对结果有影响吗,我能否直接把dice生成的文件里对应得行列直接删掉。 - 我发现我在之前的转换代码使用的时候
‘/home/Alex/Wu2017_RegistrationFusion/bin/final_warps_FS5.3/allSub_fsaverage_to
_FSL_MNI152_FS4.5.0_RF_ANTs_avgMapping.prop.mat’, …
‘/home/Alex/Wu2017_RegistrationFusion/bin/liberal_cortex_masks_FS5.3/FSL_MNI152
_FS4.5.0_cortex_estimate.nii.gz’);
会出现error,显示的是左右各一个脑区的annotation value,但最后生成的文件也是83个脑区,并没有变少,这个报错的原因我没有查到,想问下是否会对后面的分析产生影响
- 我并没有去除aal2的皮下核团和小脑后再做分析,我还不太懂这个怎么做,我目前的想法是直接用120个的做分析,然后因为aal2是在aal模板上重新分割了眶额皮层,他们的皮下核团和小脑区域应该是相同的,我有aal的相关文件,后续匹配的时候(比如dice系数)把对应的区域的值直接删掉,是可以的嘛?
- 最后就是我对怎么使用dice系数的疑惑,对于这个120*82的矩阵,我可以直接使用每列的最大值对应的行作为脑区对应吗?
因为我在MRIcron肉眼比对的时候,发现Frontal_Inf_Tri_L 对应’LH_region_3’、‘LH_region_4’、‘LH_region_5’、'LH_region_6 '、‘LH_region_38’,看dice系数文件中Frontal_Inf_Tri_L 那一行,结果发现它并不是任何一列的最大值,并且这行的最大值是LH_region_5,而LH_region_5对应的最大值脑区Frontal_Mid_2_L 。
不知道我有没有表达清楚,我在想会不会是我前期对模板的处理时出了问题,麻烦Alex老师了
- 可以的,可以直接把0相关的行列删掉。
- 我印象中最后是会有个什么error信息,但是应该是正常的输出,并不是指计算过程有错误。
- 可以的。你如果会用matlab或者python读写nii格式的数据,最简单的就是找到aal2皮下核团和小脑的编号,把这些编号直接赋值为0就可以了。
- 我明白你的意思,应该是一个双向对应的问题。比如
Frontal_Inf_Tri_L与所有annot脑区最大的对应值是LH_region_5,而LH_region_5与所有aal2脑区最大的对应值是Frontal_Mid_2_L。理想情况下,应该是双向对应的。我觉得应该是从annot脑区出发,对每一个annot脑区,去找aal2中重叠最大的脑区。如果出现两个annot脑区对应到一个aal2脑区,这个时候可以考虑如果一个aal2脑区已经被分配了,就从剩下的脑区里找。不过这种情况的出现,可能暗示着这个annot分区和aal2分区还是有一些偏差的的。根据annot分区对应的参考文献,这个annot分区是经过了一些手工编辑的,不然aal2投射到皮层会有一些问题。