Alex老师晚上好,非常感谢老师对我每一条的回复,不好意思我今天才登陆论坛回复您的消息,我刚结束期末放假啦 ![]()
![]()
![]() 然后我将对比也做完了,目前有几个小疑惑,麻烦老师了
然后我将对比也做完了,目前有几个小疑惑,麻烦老师了 ![]()
![]()
![]()
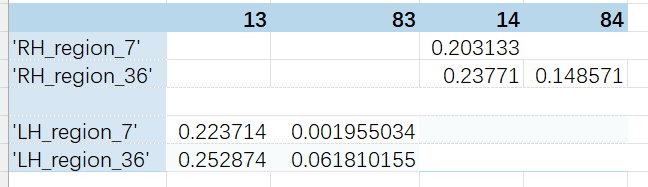
第一个是,我对比到最后,发现还剩两对脑区【Rolandic_Oper的左右脑区(13,14),Heschl的左右脑区(83 ,84)
这个图的纵列是annot对应的脑区,横列是aal2的,中间的数值是dice系数。
如果按照重叠系数进行分配,Heschl将无法匹配,所有我将Rolandic_Oper分给了annot中的脑区7,把Heschl分给了annot中的脑区36,应该是没问题的吧,我想问一下为什么会出现这种情况呀,我看’LH_region_7’对Heschl的dice系数只有0.0618,感觉占比其实是比较小的 ![]()
![]()
![]()
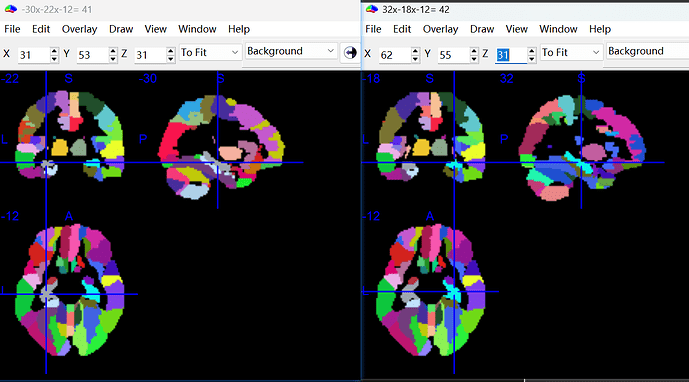
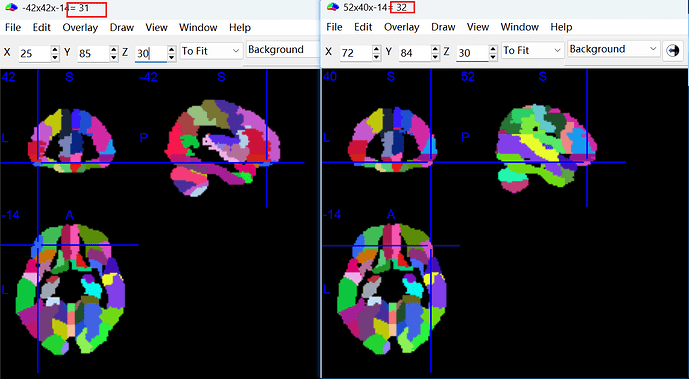
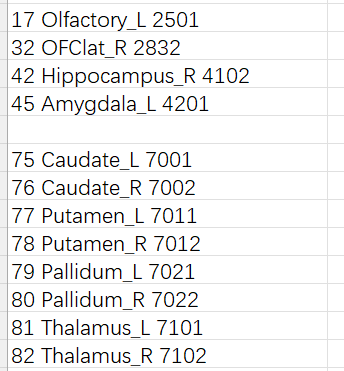
第二个问题就是我注意到有四对脑区只匹配到了左或右其中一个脑区的情况,分别缺失的是Olfactory_L、OFClat_R、Hippocampus_R、 Amygdala_L,我使用MRIcron查看后(Hippocampus和OFClat)感觉这几块区域也挺大的,不太明白为什么会少这4个脑区 ![]()
![]()
![]()