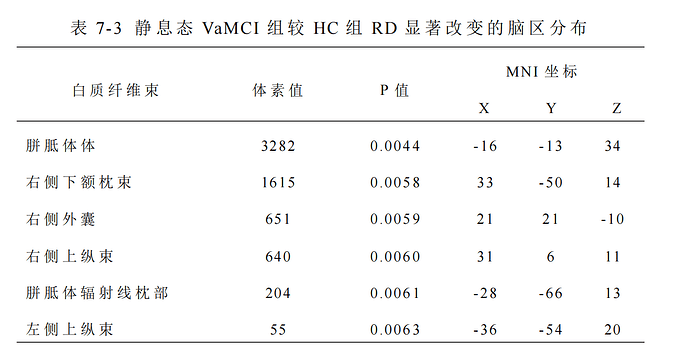
大家好,我已经分别比较3组患者与1个对照组之间做了VBM分析(模板ALL3),后续想要分析相应脑区白质纤维的差异。拟得到的结果是分析灰质损伤的脑区对应的脑区白质也存在损伤。例如:组1与对照组,vbm分析发现海马灰质体积萎缩,TBSS分析得到与海马相关的白质出现了相关损伤。
目前,TBSS分析结果里面显示 panda得出来cluster的数量很少,只有1-3个,但是fslview图片里面显示的纤维束却很多,结果显示得要比 panda里面得出来的cluster的结果好。
目前遇到的问题是
目前使用的模板是默认模板,一个WM Label Atlas:FMRlB58、一个是WM Probtract Atlas:JHU lCBM tracts maxprob thr25 1mm,请问Label和Probtract这俩模板,都是做什么的,对结果有影响。因为FMRlB58 是50个ROI、JHU 是20ROI,是否都可以用ALL3或者FMRlB58模板,这样会不会让差异的脑区种类多一些?
这个是Cluster
这个是FSLVIEW的显示结果
属于图像分析小白,基础问题比较多,希望大家不吝赐教