zhu
1
alex,
谢谢你上次关于T1图像质量的回答,这次的问题也很细节,可能你都不会注意,因为本人初次进行处理和研究,敬请包涵。
问题如下:
现在全部的步骤跑完,想拿影像指标与量表做相关,在thickness这个指标上,出现差异脑区,比如脑岛(insula),它最终cluster summary文件里只有annotation只写了insula,而我从stats文件夹里提取的70多个脑区(我是按照默认模板跑的,没有更替模板),单单脑岛它又分出了许多亚区,我接下来是不是把所有亚区的指标和量表做相关嘛?还是怎么样?烦请指教。
Alex
2
我如果没理解错的话,你就是做完了皮层指标和量表的相关,有一些显著结果。如果这是你的研究目的,那么这个分析就到此结束了。annotation只是告诉我们显著的脑区大概是什么位置。
1 个赞
zhu
3
相关还没做,差异脑区目前出来了,正如你说的annotation告诉个大概位置,但是我提取某个脑区的值很多,比如说脑岛,这个位置,又分割了一些亚区,我是都要拿来做相关吗?
Alex
4
哦哦,明白了。你先做的组间比较,组间比较有显著的差异。接下来想做显著的区域与量表的相关分析。这也是常规的分析思路。
因为你做的是vertex-wise的分析,所以一般是提取有显著组间差异的mask,提取mask的均值,然后和量表进行相关。
你也可以分析显著的团块包含的脑区,然后用脑区的均值去做相关,但这种做法是不常见的,因为显著的团块通常不包含整个脑区,或者显著的团块包含好几个脑区。
1 个赞
zhu
5
好的好的,谢谢alex!
我对你回复的话理解如下:
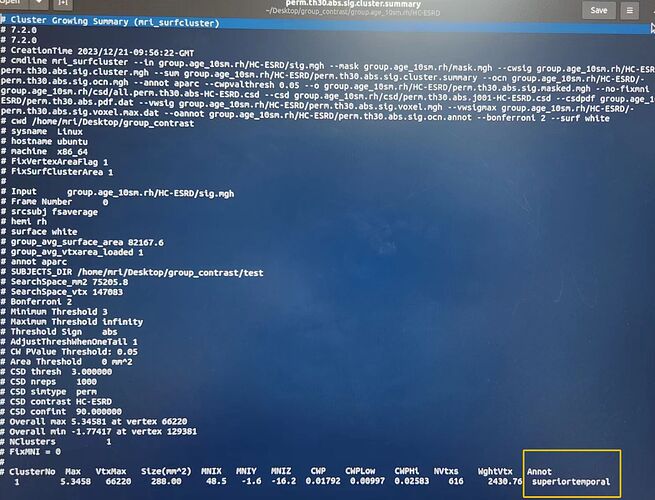
1.cluster.summary文件里汇报的只是大概的显著差异脑区,可能包含其他脑区或者只包含该脑区的一部分;
2.所以,严格来说我不能直接用从stats文件里提取的差异脑区数值来做相关,因为不准确!!!
3.所以我应该先制作label或者mask files , 然后根据label files获取关于label stats文件,从而获取差异脑区的值。(如图所示)
4.最后,拿这些差异脑区的值和量表进行相关分析!
所以,我做完了逐顶点组间分析,找到了大致差异脑区,目前先需要制作label(或者说ROI),然后提值,做相关,我的理解不知道有没有错误?
Alex
6
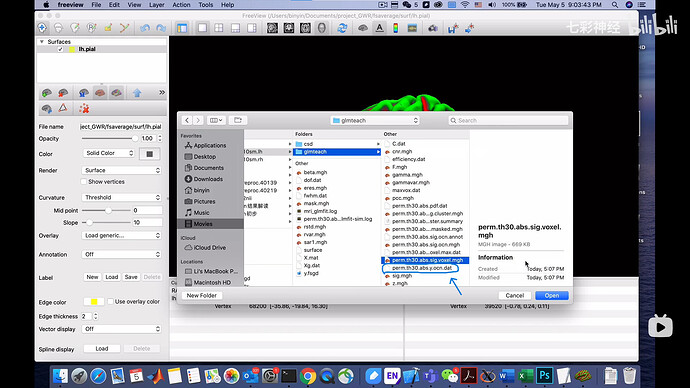
你的理解没有错,你提到的方法也是可以的。我刚去查了一下,你得到的结果里应该已经有了每个cluster的均值(应该是*sig.y.ocn.dat文件),所以不用自己再次提取了(我很久不做这种分析了,所以前面没想起来)。
zhu
7
好的,感谢alex!!!
我之后提取图中箭头所指文件的cluster均值即可!
再次感谢,小白问题较多,多有打扰了,真的很感谢,组里就我一个做结构,实在感谢!
借此提问一个问题:
如果用CAT计算皮质厚度指标,然后也想提取某一个差异cluster下所有被试的均值,应该怎么操作呢?
Alex
10
这个问题我应该在SPM的邮件列表里问过或者看到过,我在CAT12.6中测试是成功的,但是后来在CAT12.8.1中测试又不行了。具体步骤:
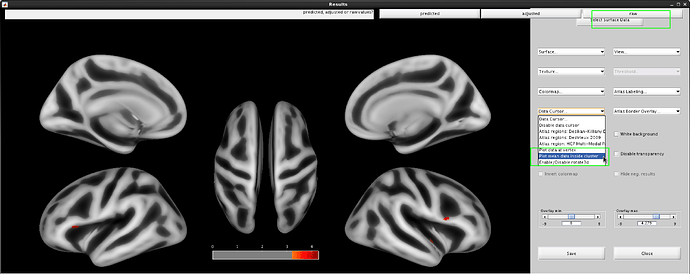
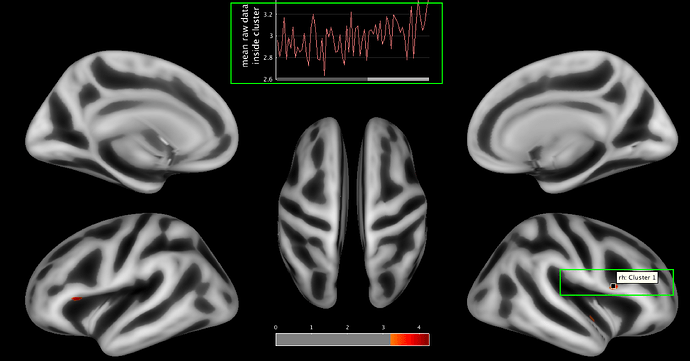
(1)打开CAT12的Display Results → Display Surface Results模块,在Select Surface Data下选择皮层分析的统计结果(卡过阈值或者转换成logP),在Data Cursor下选择plot data inside cluster,在顶部会弹出一个选项,选择Raw(另外两个选项是predicted/adjusted),表示原始的皮层厚度。
(2)使用鼠标选择一个显著脑区的位置,会在顶部显示所有被试在这个脑区的平均皮层厚度的图,同时在MATLAB环境中生成一个名为y的变量,在命令行窗口输入y,即可看到每个被试在这个脑区的平均皮层厚度的具体数值。

可以! 我用的好像是12.8.2能行
谢谢老师,get到一项新技能!操作还挺方便的
zhu
12
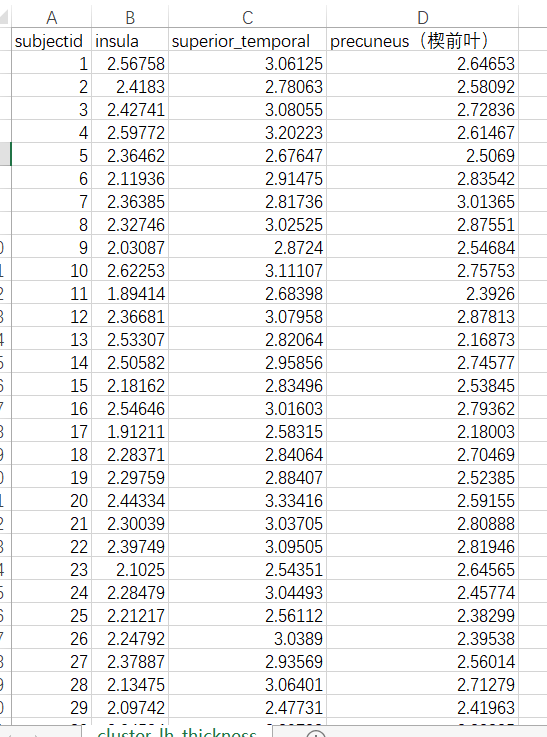
哈哈,alex,我也借此问个基础问题:我现在已经提取出来均值,但是它是全部的被试(健康组+病人组),我想拆分开来,分别做进一步分析,
想问一下图中的值的排列是否与FSGD file 里的被试排序相同?(图中的表头和和序号是自己加上的)
请问老师,CAT12计算得到的gii文件还能使用什么软件打开吗?
我把有差异的cluster保存成gii文件(好像是不可选,直接就是gii),有工具可以把它转成Nii文件吗?
Alex
16
据我所知,GIFTI是皮层格式,surface到volume的转换不是简单的格式转换。你想转成nii做什么呢?
Alex
18
我刚去SPM邮件列表搜索了一下,有两个相关的讨论,但是似乎都无法解决你的问题:
https://www.jiscmail.ac.uk/cgi-b … mp;D=0&P=219252
https://www.jiscmail.ac.uk/cgi-b … mp;D=0&P=131422
其中CAT12作者也参与了讨论,说明至少在提问的时间里CAT是无法实现这个功能的。这也是为什么,我一般皮层分析都会用FreeSurfer,这个功能用FreeSurfer就是非常简单的。
如果是我遇到这个问题,我可能有两个解决方案,一是去SPM邮件列表提问或者给CAT12作者写邮件,也许在2024年CAT12已经提供解决办法了;二是看具体分析目标是啥,选择一些迂回路线,比如你想用结构分析得到的脑区去做功能像分析的ROI,可以把功能像数据映射到皮层上分析,这个CAT是提供了工具的。又或者你想做机器学习,可以直接把脑区的值保存成.mat文件,这个CAT也是提供了相关功能。
serene
20
老师,VBM的结果能这样提取吗,我看导入VBM结果的时候 data cursor没有plot data inside cluster选项