本文尝试澄清“MNI空间”这个概念,我的理解不一定对,欢迎指正。
一、什么是空间?
在MRI脑成像里,空间(space)指的是大脑解剖结构(brain anatomy),一般通过一个三维(笛卡尔)坐标系统来描述,确定好原点的位置和三条相互垂直的轴线,这样我们就可以用坐标(X, Y, Z)来描述空间任意一个点,每一个坐标点表示的就是不同的解剖位置。
二、空间标准化
每个人的大脑在大小和形状(解剖特征)上都不相同,所以每个人的大脑空间都不一样,这个空间一般称为本地空间(native space)或者个体空间。为了使不同人的大脑(用MRI图像来表征)可以相互比较,需要将大脑(图像)从个体空间转换到一个标准空间,这个过程称为空间标准化(spatial normalization或spatial standardization)。
三、MNI标准空间
MNI空间就是一个人为定义的标准空间,这个空间的坐标系原点在前连合(anterior commissure),X轴表示左右,从左到右(Right)为正方向,Y轴表示前后,从后到前(Anterior)表示正方向,Z轴表示上下,从下到上(Superior)为正方向,这种坐标系也称为RAS坐标系。
MNI空间和MNI模板(template)几乎是同义词,通常MNI模板是T1(加权)图像,因为T1图像可以反映大脑解剖结构。在空间标准化的过程中,也是需要依靠模板来实现个体空间和标准空间的转换。
四、常用的MNI模板
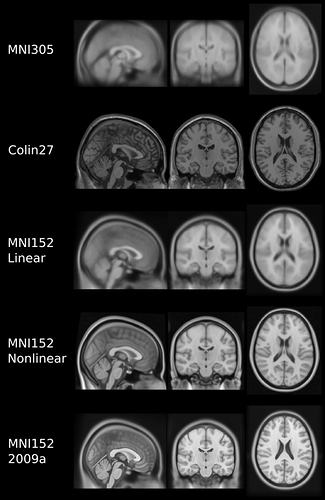
容易造成混淆的地方在于,实际上存在不同的MNI模板,也即存在不同的MNI空间。下面列举一些常用的MNI模板:
MNI305:将305名被试的T1像线性配准到Talairach空间得到的平均图像;
Colin27: 一名被试(Colin Holmes)扫描了27次T1像,将27个T1像线性配准到MNI305空间得到的平均图像;
MNI152-Linear: 将152名被试的T1图像线性配准到MNI305空间得到的平均图像;
MNI152-Nonlinear (6th-generation): 将152名被试的T1图像非线性配准到MNI305得到的平均图像;
MNI152-2009:将152名被试的T1图像非线性配准到MNI152-Linear得到的平均图像。
从上图可以看到,虽然这些模板(空间)都可以称之为MNI模板(空间),但是它们所反映的解剖特征是不同的(但是相似的),也就是说同一个坐标在不同MNI空间中表示的是不同的解剖位置。
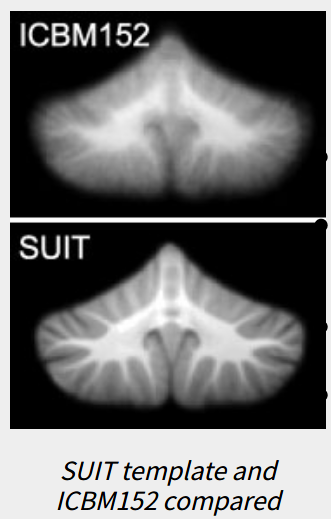
此外,还有一些容易让人误解的地方,比如Colin27在MRIcron中叫ch2,在SPM12中叫single_subj_T1。在FSL中默认使用的是MNI152-Nonlinear,但是是非对称的版本,和MNI官网给出的版本是不同的。在SPM12中使用的TPM模板实际上并不是MNI模板,而是根据549名被试生成的,虽然跟MNI152-Linear很接近,但是还是不同的。MNI152-2009有三个不同的版本(a/b/c),在构造方法上有差别。在CAT12的不同版本中使用的是不同的模板,但是都不是MNI空间的,早期版本(比如12.5)接近MNI152-Nonlinear,近期版本(比如12.8)接近MNI152-2009。
五、分区模板
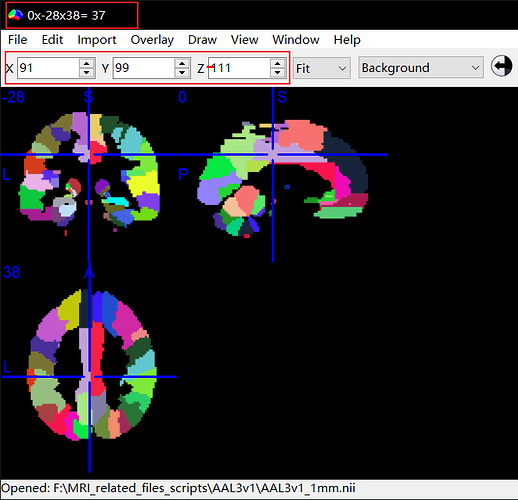
和MNI空间/模板相关的一个概念是分区地图(parcellation atlas)。分区地图指的是对图像进行结构或功能的划分,比如标记出哪些位置是海马。之所以会和MNI空间有关系,是因为分区地图也存在位于哪个空间的问题。比如AAL分区地图是对Colin27进行划分的,所以AAL是位于Colin27空间的。如果我们把AAL用于位于MNI152-Nonlinear空间的图像,那么误差就会很大。这是我们在使用分区地图进行数据提取或者脑区定位的时候需要考虑到的因素。
六、参考资料
https://www.lead-dbs.org/about-the-mni-spaces/
https://nist.mni.mcgill.ca/atlases/
(本文部分内容最初于2019年发布在个人公众号上,这里重新进行了整理,并补充了一些内容。)