利用freesurfer的GLM以及多种比较校正搜索差异化脑区。
对6个组别被试进行探究,有基线和随访两个时间点。
(1)在6个组别内对两个时间点之间进行分析,有较多脑区存在差异(聚类结果);
(2)对随访时间点所有被试数据进行分析,不同组别之间也有较多脑区有显著差异(聚类结果)。
问题:
前面的分析基于aparc(68结构分区)作为annotation,但我改成aparc.a2009s(148分区)后聚类不出什么好结果,这是什么原因呢?有什么改进的建议呢?这种会是什么步骤出了问题呢?
大神们求解!不胜感激。
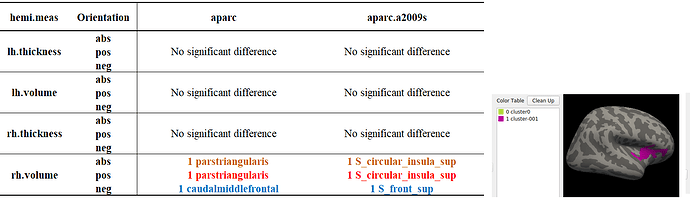
你这里所说的“聚类结果”是什么意思?你的意思是不是用68个分区有显著差异脑区,用148个分区没有显著差异脑区了?
对的对的,聚类结果就是通过多重比较校正之后还有显著差异的区域,用了aparc.a2009s以后在不同比较方案几乎都没什么显著差异
你分析的方法是,先根据分区模板提取出不同脑区的指标,比如68个脑区的平均皮层厚度,然后在每个脑区分别进行统计分析,最后对所有脑区结果进行多重比较校正,是这样的思路吗?
预处理和glmfit命令不会对特定脑区模板的那些脑区进行分区统计,是逐顶点对整个图像进行统计,在glm-sim多重比较校正聚类分析的时候才会考虑脑区(option可选择选择annot)
如果你是做的vertex-wise的统计分析,那么改变annot应该不会影响结果的,annot只是用于标记显著cluster的脑区标签。你切换annot前后,cluster数量和P值会变化吗?
summary中annot显示的是差异最大的vertex的脑区,而不是整个cluster涉及到的所有脑区。
原来如此!针对这个情况,我还有两个问题想请教一下您:
(1)如果想获得cluster涉及的多个脑区,是只能通过人为检查的方法吗,还是有什么方法可以快速获得相应的所有脑区信息呢。
(2)如果在mri_glmfit分析的时候我将此前发现的相关所有脑区作为mask输入,会对研究结果有好处吗?
- 我确实不清楚FreeSurfer是否提供了相关的命令,如果自己编程实现的话,根据cluster文件和分区annot文件应该可以提取每个cluster包含的脑区,比如提取显著脑区对应的vertex number,然后根据vertex number去annot文件里查询脑区标签。
- 有可能有好处,因为缩小了搜索范围,多重比较校正更宽松了。但是如果确实没有差异,校正再宽松也没用。
1 个赞
谢谢您,我暂时没什么问题了,我再去多多尝试一下 ![]()