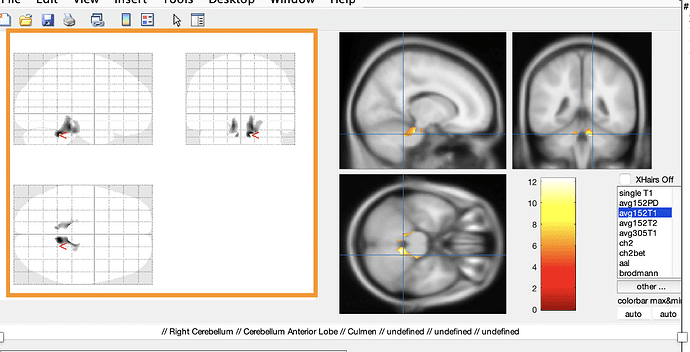
各位老师好,我之前利用VBM(cat12下)处理比较受试者和健康者之间gray matter之间的区别,得到了一些与文献相同的脑区的区别(图1);
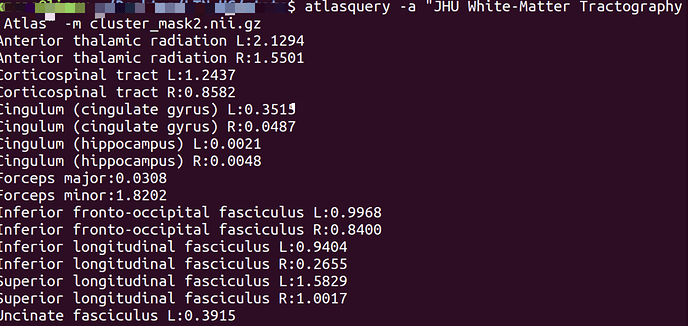
其次,利用TBSS 基于voxelwise下,进行FWE校正后得到的白质部分(图2)
我想请教下各位老师方法论上的问题,我想将这两部分的比较联系起来,是否需要借助结构态的网络才可以实现?或者有别的方法吗?
查阅了相关文献:1.结合 gray matter and white matter, 文章中跟之前两步一样,但没有进行联系,往往是各自说出对应的结果;
2.查阅结构态的脑网络,是在FA图像的基础上,利用全部脑区的节点或者ROI进行分析,从而得到脑区之间联系变化——这部分结果与TBSS 白质得到的内容相似,出现例如左侧上纵束纤维数目减少,从而得到脑网络之间数值,但这个与灰质全脑比较的结果没有联系。
各位老师,我的想法具有可实现性吗?如果可以的话,还需要掌握学习哪些技术流程呢?我之前的思路有也可能误区,请大家指点,比心!
Alex
2
我以前也想过这个问题,但是没有好好去查查文献。我自己的做法就比较简单,就是算一下GM和FA cluster之间的相关,这样可以说明灰质的个体变化与白质的个体变化是关联的,类似与GM和量表的关联分析。另外,可以看看GM区域和白质纤维束连接的脑区是否有重叠,这样也可以解释两者之间的潜在关系。
也可以在GM结果基础上去做分析,比如用GM cluster做seed去做纤维束追踪(根据DWI数据),看看追踪得到的纤维束是否和FA结果涉及的纤维束有关系。
好的,谢谢老师,我试一下您说的方法。
还有一个问题是,vbm处理时,也可以换为白质所在的序列,这个结果会对最后的显示有帮助吗?我试了后感觉联系不是很明显
再次感谢老师!
不好意思,cat12分割为灰质,白质,脑脊液,vbm组间对比时,将序列换成白质后,跟使用灰质序列对比没有明显的区别,不知道是不是存在别的错误
Alex
6
你的意思是,你也做了白质体积的VBM分析,是吗?我不太清楚“没有明显的区别”是什么意思?
对的,我也做了白质体积的VBM的分析,没有明显区别的意思是:对比之前灰质VBM处理后体积减少的脑区,白质体积也出现了减少(cerebeiium thalamus等),还有在运行框出现了gray matter的脑区报告(白质VBM)。很疑惑这些是不是正确的,还是因为矫正的误差导致的。
Alex
8
感觉不太对,你可以直接把灰质和白质的VBM结果重叠起来看看两者之间的关系是什么。虽然报告的是同一个区域比如Cerebellum,但是实际位置不一样,比如相邻的区域。可能跟用来报告脑区的分区模板的精度也有关系。你在用CAT做统计分析的时候,可以尝试把阈值调高一些,把比较限制在深部白质,避免在灰白质交界处出现结果。
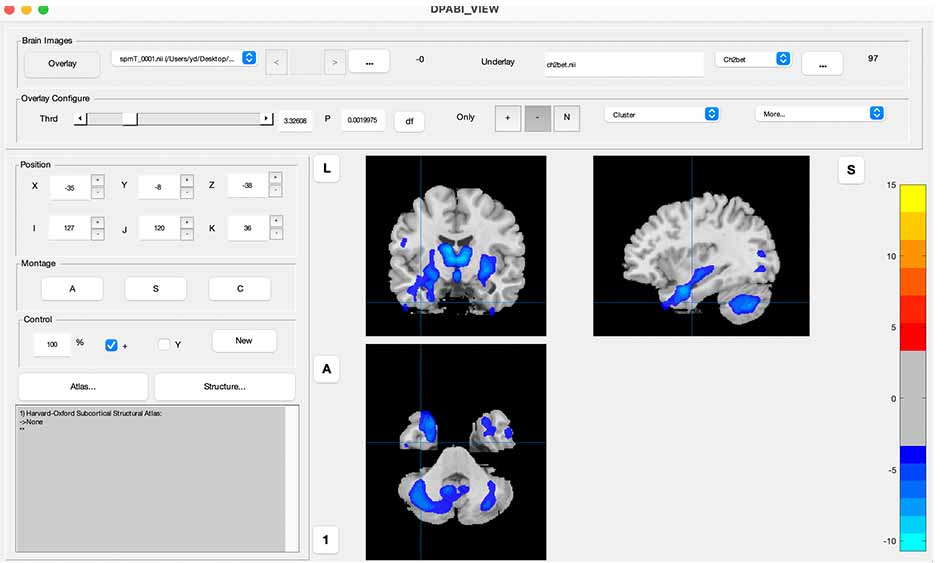
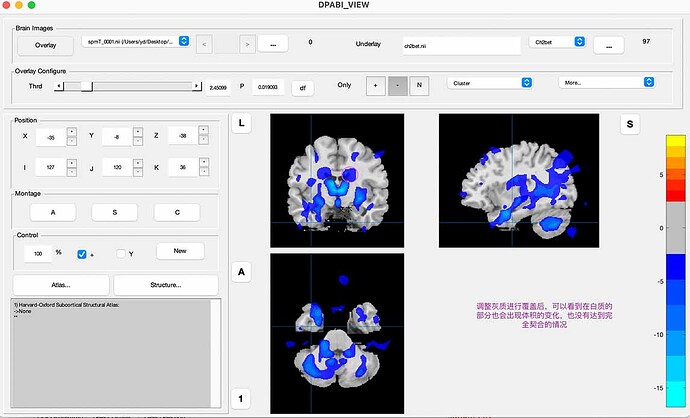
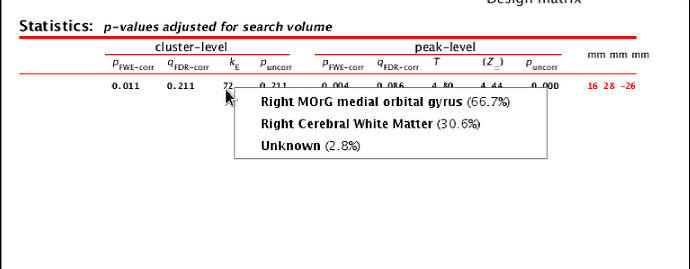
谢谢老师,我根据你的建议,用dpabi viwer 的部分进行了展示,白质和灰质部分进行了叠加(图1-图4),有部分是重复的,但绝大多数是独立的,但也不是完全互补的,之前使用的是xjview报告cluster的,结果比较多,我翻看您之前给其他人的解答,您一般不用xjview进行结果的展示,我也查询了原作者的一些问题解答(图5),以及其他人使用xjview出现的问题和解答(图6),所以我觉得我出现相同脑区的原因也有一部分是因为单纯依赖xjview的结果了
提出这个问题的原意是因为,对于灰质VBM报告的团块结果不太理解,完全依赖xjview,所以很多东西我找不到解释依据,所以想通过宏观的白质和微观的白质进行联系
后来我改进了下;在threshold那里设置到了0.01,结果比之前设置的好一些,交界区的东西少了很多(我是用spm进行分析的),我想了想,跟几个因素有关系:1.在设置阈值的时候,threshold的选择,2.对于团块的大小确定;3.FDR还是FEW校正;根据既定解剖知识,灰质是功能区的部分,白质作为纤维连接部分。我又接着查阅了相关白质VBM的文献,发现最后还是白质团块最后还是作为连接部分与疾病的机理进行讨论的。很感谢老师,使我对问题进行了复盘。
想问问老师,不知道您对cluster报告的结果 有无更好的建议呢?
Alex
11
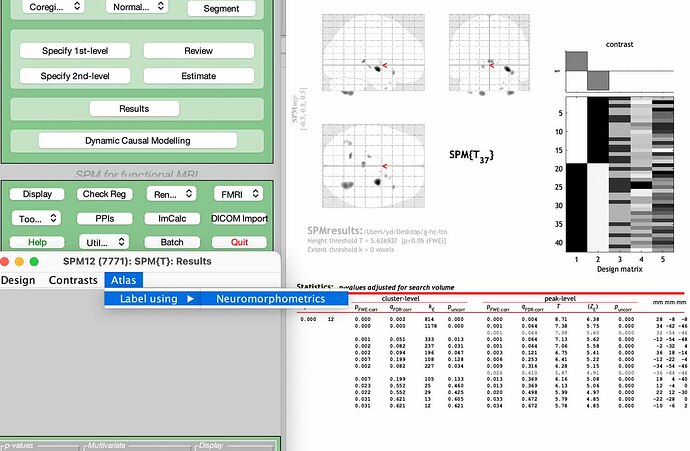
(1)你使用的阈值(0.01)可能还是太低了,所以有很多边缘的地方也纳入了分析。CAT12手册上推荐的是0.1,也就是至少有10%的概率为灰质或白质才纳入分析。(2)你的结果这么多,做了多重比较校正吗?(3)如果是有显著结果,报告结果的话,我一般使用SPM12 Results界面的Atlas功能(见下图)。这个功能在CAT12或者SPM12的使用手册里有介绍。
好的,老师,我调整至0.1后还是有着较多的差别脑区,改善到到1后好了很多;进行了FWE0.05水平的校正。
我是想得到脑区对应的体素值和MNI左边点的位置,所以得借助一下dpabi viwer部分
我的SPM label using 没有那么多脑区,老师是自己手动添加的吗?
Alex
13
好吧,那你的组间差异确实挺大的。我把CAT12自带的一些分区模板也复制到SPM12下面了,所以会多一些分区。不过SPM12自带的neuromorphometrics一般也够用了。
好的,那老师,如果需要这种格式的体素以及对应的指标,是不是spm下的信息不够用了呀。
Alex
15
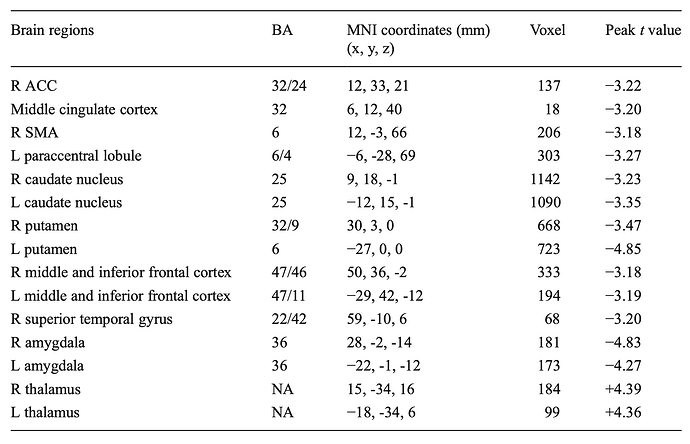
有的,KE那一列就表示Cluster size,最右边是坐标,T那一列就是T值。不过其实你用啥工具都行,这些信息应该都是一样或者非常接近的。只是脑区的名字用不同分区模板差别会比较大。
老师,我看这个表格是根据脑区分门别类整理好的,但是报告出来的每个cluster里面有重复的脑区,对应脑区的mni 还有t值,这个应该怎么实现呢?
Alex
17
你多参考一些文献,并不是都是以脑区来整理结果的,我觉得更自然的是以cluster为单位整理结果,因为一个cluster往往涉及好几个脑区、同一个脑区也可能出现在一个cluster里。
我想你是否在VBM分析的时候 basic model 处没有加上阈值,导致交界区有差异,我看文献一般会设置灰质绝对值是0.1或0.2,或者用一个组水平的mask,你可以试试。