您好,我想在一个被试的个体空间将丘脑亚区分割出来,以便于清除选择丘脑某个亚区作为靶点。是使用FSL直接将使用AAL3图谱配准到个体空间可以吗?这样准确吗?使用freesurfer怎么操作?有没有FS空间的丘脑亚区分割图谱啊?谢谢!
您好,我FSL使用以下代码
bet2 T1.nii.gz T1_brain -f 0.5 -m
flirt -ref /home/liubin/fsl/data/standard/MNI152_T1_2mm_brain -in T1_brain.nii.gz -omat T1toMNI.mat
fnirt --in=T1_brain.nii.gz --aff=T1toMNI.mat --cout=T1toMNI_cwarp.nii.gz --config=T1_2_MNI152_2mm
invwarp -w T1toMNI_cwarp.nii.gz -o MNItoT1_cwarp -r T1_brain.nii.gz
applywarp -i AAL3v1_1mm.nii.gz -r T1_brain.nii.gz -o T1_AAL3.nii.gz --interp=nn
可以配准出来的图像偏移是怎么回事?
谢谢
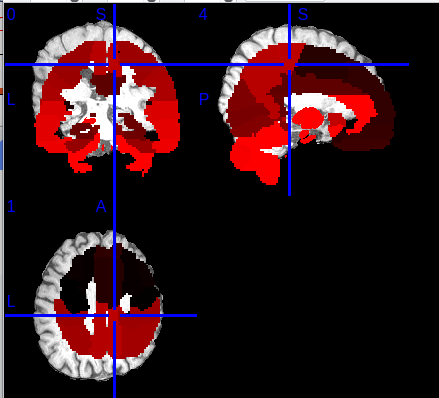
直接配准是可以的,但是可能不够精确。FS有分割丘脑核团的模块:https://freesurfer.net/fswiki/ThalamicNuclei,虽然FS也不一定精确,但是FS认可度比较高。就跟海马亚区一样,我个人觉得常规分辨率T1数据分割结果都是不够精确的。
(1)你应该检查从个体T1配准到MNI的结果是否准确,再考虑把AAL模板转换到个体空间。
(2)用fnirt的时候应该使用没有去颅骨的图像(即T1.nii.gz),可以仔细看看fnirt的wiki。
(3)建议试试ANTs,配准效果比fnirt要好很多,而且更稳定(fnirt和bet是我感觉FSL里表现最差的软件)。
太感谢了!!我就觉得FS应该有像海马亚区分割的功能,谢谢!!!
学习了,感谢~
我找到原因了,是我粗心了,applywarp后面忘了加-w了。配出来大体还行,就是比较粗糙