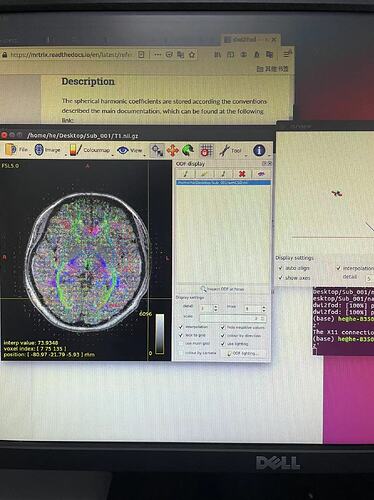
我的数据是b0+b1000的single shell的DTI数据,所以我用的命令函数如下:
dwi2response tournier input.nii out_wm.txt
dwi2fod csd input.nii out_wmCSD.nii
*注:input.nii是配准到T1空间以后的DTI图像。
把输出的wmCSD.nii叠在T1上以后,就发现有许多体素分布在颅外,如图所示
配准用到的命令是:
flirt -dof 6 -cost normmi -ref T1w.nii -in b0.nii -omat T_fsl.txt
transformconvert T_fsl.txt b0.nii T1w.nii flirt_import T_DTI2T1.txt
mrtransform -linear T_DTI2T1.txt eddy_corrected_dti.nii out_align.nii
没用过MRtrix,你对DTI数据做过mask吗?(就是把脑外的数据置0)。
另外,MRtrix的优点是啥?我看有没有必要去学一下:lol
不好意思,过了这么久才看到。后面我加了-mask限制以后,就解决了这个问题。
就我目前的使用感受来说,MRtrix3是一个集成的软件,所以可以在里面选择比较合适的算法,不局限于单个软件的算法。
做纤维追踪速度会比fsl快很多。
另外,这个软件的网站把每一条命令的用法和示例都写的很详细,开发者在论坛也很活跃。
谢谢
有详细文档和论坛的软件就很值得学一学!