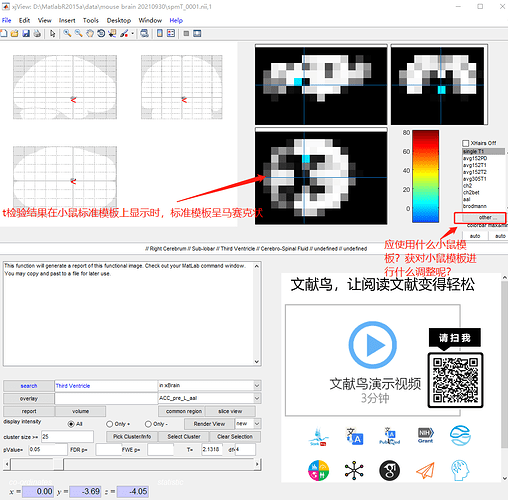
用xjview显示小鼠脑的T1像的 VBM t检验结果时,导入的鼠脑标准模板在xjview内呈马赛克状。
请问如何将xjview切换为小鼠脑的浏览模式?谢谢!!
(1)不知道你想用xjview实现什么样的目的?如果是可视化的话,可以试试其他软件(2)似乎是图像的分辨率的问题?你现在小鼠模板的分辨率是多少?
(1)想用xjview进行t检验显著区域的可视化及解剖位置分析。请问老师又其他软件推荐吗?
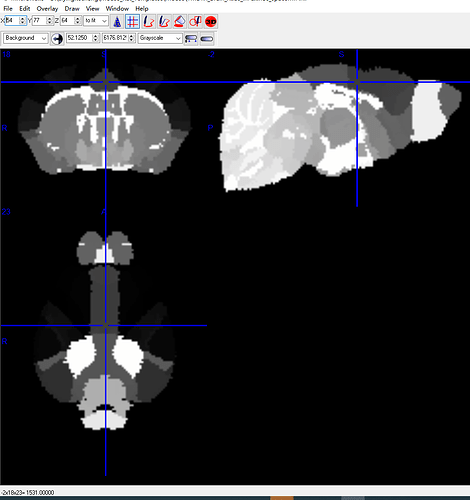
(2)小鼠模板在mricron中能正常显示。分辨率是165*230*135
解剖位置分析的话,估计xjview不行吧,因为(据我所知)xjview只有人脑的分区信息。我确实没做过小鼠数据,不知道一般都用什么软件做。如果是我的话,我可能会用mricron进行可视化,再去找一个小鼠的分区模板来标记显著区域的解剖位置。
老师您好,我有个想法,参考dpabi中对人的fMRI分析结果可视化功能,每一种图谱对应需要用到的一个是脑区图谱文件(小鼠图谱模板有相应的文件),一个是label文件。
我的想法是参考其中一个图谱(比如aal图谱)的label内容(其内包括:划分的脑区名称,脑区label,对应的坐标),把它对应修改为小鼠的label?疑问在于,这里面每个脑区对应有一个坐标,那怎么去自定义小鼠图谱中每个脑区对应的坐标呢?
如果你有一个脑区图谱文件(比如,nii格式的文件),那每个脑区的坐标(可能是用这个脑区的中心/重心坐标来表示)就应该可以获得。不过我其实没太看懂你的问题,可能是因为我并没有用dpabi做过可视化,不清楚实现原理。
我去SPM的邮件列表里搜了一下,里面给出了一种代码实现的方法:https://www.jiscmail.ac.uk/cgi-b … p;L=SPM&P=R4324
如果你用FSL的话,可以用cluster命令获得一个脑区的中心坐标。
收到,非常感谢!
你好,目前我也在处理小鼠的数据,希望可以和你一起交流一下,我已经私信你我的联系方式